Genetické testování – polygenní skóre
Genetic testing – polygenic score
In recent years, efforts to incorporate genetic dispositions into clinical practice have led to the development of genetic risk scores (GRS). These should summarise the complex genetic risk as a single value derived from the results of testing a dozens to thousands of individual polymorphisms. There are several types of genetic risk scores. The unweighted GRS uses the simple sum of the risky alleles present, the weighted GRS takes into account the relative effect of individual variants on the phenotype (OR, HR or β coefficient), and finally the polygenic risk score (which includes up to hundreds of thousands of polymorphisms) also takes into account the linkage between the individual alleles. A number of studies have confirmed the significant effect of GRS on the estimation of disease risk or prognosis. Because studies are highly heterogeneous in the number and selection of polymorphisms included in GRS calculations, it is not possible to determine what nominal value already represent a clinical risk. There is also a significant lack of studies looking at interactions between GRS and lifestyle. GRS analyses represent a next step for the use of genetic information in everyday, primarily preventive, clinical diagnosis.
Keywords:
cardio-vascular disease – genetic – genetic risk score – prediction
Autoři:
Jaroslav A. Hubáček 1,2
Působiště autorů:
Centrum experimentální medicíny, IKEM, Praha
1; III. interní klinika – endokrinologie a metabolismu 1. LF UK a VFN v Praze
2
Vyšlo v časopise:
AtheroRev 2023; 8(2): 95-101
Kategorie:
Přehledové práce
Souhrn
Snaha zahrnout do klinické praxe genetické dispozice ústí v posledních letech k vytváření genetických rizikových skóre (GRS). Ta by měla shrnout komplexní genetické riziko jedinou hodnotou získanou z výsledků testování řady (desítek až tisíců) individuálních polymorfizmů. Existuje několik typů genetických skóre. Nevážené GRS využívá prostý součet přítomných rizikových alel, vážené GRS bere v potaz relativní vliv jednotlivých variant na fenotyp (OR, HR nebo β-koeficient) a konečně polygenní skóre, zahrnující až statisíce polymorfizmů, bere v úvahu i provázanost výskytu jednotlivých alel. Celá řada studií potvrdila důležitost GRS v odhadu rizika onemocnění nebo jeho prognózy. Protože studie jsou značně heterogenní co se týká počtu a výběru polymorfizmů zahrnutých do výpočtů GRS, nelze určit, jaká nominální hodnota je již riziková. Rovněž prozatím citelně chybí studie sledující interakce mezi GRS a životním stylem. Analýzy GRS představují další posun k využití genetických informací v každodenní, především klinické diagnostice v rámci primární prevence.
Klíčová slova:
genetika – genetické rizikové skóre – kardiovaskulární onemocnění – predikce
Úvod
Úloha genů v determinaci civilizačních onemocnění
Analýza genetické variability dává možnost opravdu včasné detekce jedinců ohrožených kardiovaskulárními onemocněními (KVO). Přítomnost alel predisponujících k civilizačním onemocněním (a tedy i KVO) lze zjistit v jakémkoli věku a získání alespoň hrubé sekvence lidského genomu [1] umožnilo zkompletovat jeho variabilitu a rozpoznat běžně se vyskytující rizikové alely.
Civilizační onemocnění, KVO nevyjímaje, jsou ze své podstaty polygenní, tj. na jejich vzniku se spolupodílejí varianty řady genů. Jejich vliv na fenotyp je obvykle považován za „malý a nevýznamný“, nicméně individuální varianty v genu pro ANRIL (regulační nekódující RNA) zvyšují riziko KVO o cca 70 % [2], transkripční faktor TCF7L2 zvyšuje riziko diabetes mellitus 2. typu (DM2T) přibližně 2násobně [3] a jeden FTO-polymorfizmus navýší tělesnou hmotnost až o 3 kilogramy [4]. Většina potenciálně zajímavých variant pak ovlivní fenotyp/riziko KVO o 5–40 %, což je plně srovnatelné s rizikem spojeným s vyšší hladinou cholesterolu [5] nebo s obezitou [6].
Odhlédneme-li od rizikových faktorů životního stylu, civilizační onemocnění jsou v populaci častá proto, že existují stovky běžně se vyskytujících rizikových alel a každý jedinec jich tedy má relativně vysoký počet. To je podporováno i faktem, že často je riziková alela ta četnější.
Do určitého počtu jsme schopni přítomnost rizikových alel tolerovat, ale jejich přílišná akumulace vede ke „zhroucení systému“ a podporuje propuknutí nemoci.
Snaha zjistit toto kritické rizikové množství negativních alel a vyjádřit komplexní genetické riziko jedinou, obecně porovnatelnou a snáze uchopitelnou hodnotou, vyústila v sestavování a analýzy genetických rizikových skóre.
Genetické rizikové skóre
Genetické skórování zažívá v posledních letech boom, přestože první práce na toto téma vznikly již před čtvrt stoletím [7,8]. Základní principy tohoto skórování byly v časopise AtheroReview stručně zmíněny před několika lety [9].
V literatuře lze narazit na pojmy nevážené genetické rizikové skóre (uGRS – unweighted Genetic Risk Score), vážené genetické rizikové skóre (wGRS – weighted Genetic Risk Score) a polygenní rizikové skóre (PRS – Polygenic Risk Score). Nelze je mezi sebou zaměňovat, každé skóre je přesně definované [10–12].
Nicméně striktní požadavky na to, kolik polymorfizmů je vhodné analyzovat a sumarizovat, stejně tak kdy je již riziko spojené s jednotlivou alelou dostatečné k zahrnutí do skóre a jak vysoké skóre představuje riziko rozvoje onemocnění, neexistují a prozatím je toto ponecháno na invenci výzkumníků.
Nevážené genetické rizikové skóre
Nevážené genetické skóre (uGRS) je prostým součtem všech přítomných rizikových alel (RA) pro daný fenotyp (příklad výsledku – graf). Nositelé dvou protektivních/neutrálních alel obdrží za každý analyzovaný polymorfizmus pro výpočet skóre hodnotu „0“ (žádná riziková alela), heterozygoti hodnotu „1“ (jedna přítomná riziková alela) a nositelé dvou rizikových alel hodnotu „2“.

Výhodou tohoto přístupu je jeho jednoduchost (schéma 1). Stačí pro každou variantu stanovit, která z alel je riziková. Nevýhodou může být, že hodnoty „0“ „1“ a „2“ jsou pro všechny zařazené geny/alely identické, bez ohledu na to, jak velký je vliv sledovaných variant na fenotyp. Stejnou hodnotou do uGRS tak přispějí alely zvyšující riziko onemocnění o 10 %, o 20 % nebo třeba i o 50 %, podobně pak alely navyšující plazmatický cholesterol o 0,05 mmol/l nebo o 0,25 mmol/l.
Schéma 1.1 ukazuje rozdílný počet rizikových (tmavě zelené) a neutrálních/protektivních (světle zelené) alel v případě, že
přítomnost alespoň jedné rizikové alely nese stejné riziko jako přítomnost dvou rizikových alel. uGRS tak bude pro jednotlivé
řádky 14; 11; 8 a 5.
Schéma 1.2 počítá s tím, že každá riziková alela zvyšuje pravděpodobnost onemocnění, a je tak třeba sečíst informace
z obou chromosomů. Jedinec v prvním řádku tak má hodnotu uGRS 19, ve druhém řádku pak 15.
Schéma 1.3 znázorňuje stav pro výpočet wGRS – každý individuální polymorfizmus má jinou rizikovou hodnotu vyjádřenou
velikostí kruhu (neutrální alely nejsou zobrazeny).

Vážené genetické rizikové skóre
Vážené genetické skóre výše zmíněnou rovnost odstraňuje a bere v potaz skutečný zjištěný efekt každé přítomné alely (schéma 1.3). Například varianty již zmiňovaného genu pro ANRIL tak mají při výpočtu rizika KVO vyšší váhu než ostatní. Je tedy vždy nutné nejen určit, která alela je riziková, ale i vypočítat, v jakém poměru zvyšuje riziko onemocnění (formou OR/odds ratio/poměr šancí, RR/relative risk/relativní riziko nebo HR/hazard ratio/poměr rizik). Tato hodnota je pak obvykle před součtem logaritmována.
V případě výpočtu wGRS ke kontinuálním proměnným (např. hladiny cholesterolu, výše BMI či hodnoty krevního tlaku) se vychází z hodnot β-koeficientů, které se ale již dále nelogaritmují. Použití váženého skóre je tak na první pohled správnější a mělo by mít vyšší výpovědní hodnotu.
Nicméně hodnoty OR i β-koeficientů se pro jednotlivé polymorfizmy často liší v závislosti na charakteristikách (pohlaví, věk, převažující životní styl v populaci) porovnávaných skupin. Přesná hodnota wGRS je tak platná pouze pro danou populační podskupinu/kategorii. Vyšší skóre tak dobře charakterizuje rizikového jedince, ale ani zde nelze přesné zvýšení rizika zcela zobecnit.
Polygenní rizikové skóre
Polygenní rizikové skóre počítá primárně se zařazením řádově většího počtu polymorfizmů než vážené či nevážené GRS. To s sebou přináší dva zásadní problémy. V první řadě – pokud analyzované varianty leží v řetězci DNA příliš blízko u sebe (což reálně mohou být i statisíce nukleotidů), nemusí být jejich přítomnost náhodná (jsou v různém stupni vazebné nerovnováhy – Linkage Disequilibrium – LD). Určité kombinace alel se tak vyskytují ve vzorku mnohem častěji, než by odpovídalo náhodné kombinaci – některé pak logicky méně často (podrobně schéma 2). Alely se tak dědí ve vazbě a haplotypu, a jejich efekt tedy není nezávislý. Je tak třeba znát míru provázanosti jednotlivých polymorfizmů (což vyžaduje mnohem sofistikovanější statistické analýzy) a výpočet podle toho vhodně korigovat.

Druhým problémem je, že při tak vysokém počtu zahrnutých polymorfizmů jsou efekty řady z nich velmi nízké (OR 1,01–1,05). To zvyšuje riziko, že do výpočtu budou zahrnuty alely s falešně pozitivním nálezem a při replikacích a budoucím použití v klinické praxi může být výsledek zkreslen nežádoucím směrem.
Genetické rizikové skóre a hladiny plazmatických lipidů
Informativní uGRS lze sestavit i z několika málo (např. z pouhých čtyř [13]) jednonukleotidových polymorfizmů (SNPs – Single Nucleotide Polymorphism). Hodnoty LDL-cholesterolu (LDL-C) a celkového cholesterolu (Total Cholesterol – T-C) byly v této studii vyšší o ~ 0,5 mmol/l u nositelů 8 rizikových alel (RA) než u nositelů 4 a méně RA. To je srovnatelné se zjištěními z několika studií analyzujících řádově vyšší počet genů. GRS stanovené na základě 23 SNPs bylo spojeno s hladinami T-C v rozmezí ~ 5,2 mmol/l (nositelé méně než 11 RA) a ~ 6,0 mmol/l u osob s 18 a více RA [14]. Shahid et al [15] konstruovali GRS ze 21 SNPs (z původně 361 analyzovaných) a zjistili hodnoty T-C ~ 4,7 mmol/l u nositelů 14 a méně RA zvyšujících cholesterol a ~ 5,7 mmol/l u nositelů s 21 a více RA.
Analýza dvou britských Biobank (WHII a BWHHS) [16] prokázala rozdíly mezi opačnými kvantily GRS 0,6 mmol/l pro LDL-C a 0,9 mmol/l pro T-C. Zajímavé je, že autoři použili různé (i když významně se překrývající) soubory SNPs pro stanovení T-C (20 SNPs) a LDL-C (22 SNPs) uGRS. Pozdější [17] rozsáhlá analýza identických vzorků využila výsledky z celogenomového sekvenování a PRS, zkonstruované z téměř 8 500 SNPs, vysvětlila více než 20 % rozptylu koncentrace LDL-C.
Genetická riziková skóre se intenzivně diskutují i v souvislosti s familiární hypercholestrolemií (FH). Ukázalo se, že kumulace běžných „cholesterol zvyšujících“ alel pouhých několika (6 či 12) genů vede k fenotypu zvanému polygenní FH/„pseudo FH“, který je klinicky nerozlišitelný od monogenní FH [18]. Výše zmíněná studie [17] ukázala, že efekt PRS je srovnatelný ve FH a nonFH populacích. Za zmínku rovněž stojí, že při porovnání různých PRS složených ze 165–1633 SNPs pro diagnózu polygenní hypercholesterolemie mělo nejlepší výpovědní hodnotu PRS složené z nejmenšího počtu polymorfizmů [19]. Podobně jako v souvislosti s vysokými hladinami cholesterolu, výrazná kumulace RA je pozorována i u subjektů s hypertriglyceridemií [20,21].
Genetické rizikové skóre a riziko diabetu
Z dalších tradičních rizikových faktorů aterosklerózou podmíněných KVO se analýzy GRS využívají především (i když existují studie popisující GRS determinaci i pro hypertenzi [22] či obezitu [23]) pro odhad rizika DM2T.
Výrazného zpřesnění predikce DM2T bylo dosaženo pomocí wGRS z více než 60 variant [24]. Jedinci s wGRS v nejvyšším kvintilu měli téměř 3násobné riziko vzniku DM2T oproti jedincům v nejnižším kvintilu. Výsledky ze švédské Malmö Diet and Cancer Study cohort [25] ukázaly, že přidání genetických dat zlepší predikci vzniku DM2T především u jedinců s nízkým rizikem dle životního stylu.
Existující heterogenitou GRS a rizik DM2T se podrobně zabývala práce Godarziho et al [26] provedená na Američanech mexického původu. 500 SNPs bylo rozděleno dle svých funkcí do 4 různých, početně heterogenních a částečně se překrývajících skupin (např. skupina B-buňky, skupina inzulinová rezistence a další). Zajímavé je, že většina analyzovaných SNPs spadala do kategorií „funkce nikdy nezkoumána“ a „neklasifikováno“. Nejvyšší predikční síla byla spojena s GRS zkonstruovanými pomocí celého souboru SNPs. Z jednotlivých podskupin měla nejvyšší výpovědní hodnotu skupina SNPs ovlivňujících funkci B-buněk. Naproti tomu skóre pro skupinu SNPs „inzulinová rezistence“ nemělo žádnou souvislost s DM2T.
Genetické rizikové skóre a riziko KVO
Výsledky prakticky všech relevantních, dostatečně početných a výpovědních studií sledujících GRS a riziko KVO byly shrnuty v práci kolektivu Yun et al [27] z loňského roku. Některé z nich je vhodné pro představu a porovnání stručně zmínit.
Analýza UK Biobanky ukázala [28], že GRS je užitečný nástroj k detekci jedinců ohrožených KVO primárně v mladším věku (muži do 40 a ženy do 45 let).
Khera et al [29] analyzovali extrémních 6,6 milionu SNPs pro kvantifikaci rizika KVO, přičemž 5 % osob s nejvyšším GRS mělo „pouze“ 3,7krát vyšší riziko časného infarktu myokardu ve srovnání s ostatními jedinci. Jen o něco nižší OR (2,96) bylo přitom dosaženo při použití GRS založeného na pouhých 21 SNPs [15]. Dánská studie při použití 45 SNPs oproti tomu zaznamenala sice významné, nicméně marginálně zvýšené riziko infarktu (o 5 %/jeden bod GRS) [30].
Nejasnosti a další směry výzkumu v oblasti GRS
Je třeba zmínit, že řada i poměrně rozsáhlých studií nenalezla významný vliv GRS v předpovědi onemocnění, nebo nezlepšila předpovědi jejich vývoje založené na tradičních rizikových faktorech [31–33]. Příčin může být celá řada. Často se jedná o skutečnost, že do skórování jsou primárně zařazeny geny ovlivňující právě ony „tradiční“ rizikové parametry a touto duplikací je GRS v aditivním modelu diskriminováno.
Některé body znesnadňující interpretace a klinické aplikace jsou stručně shrnuty v tab.
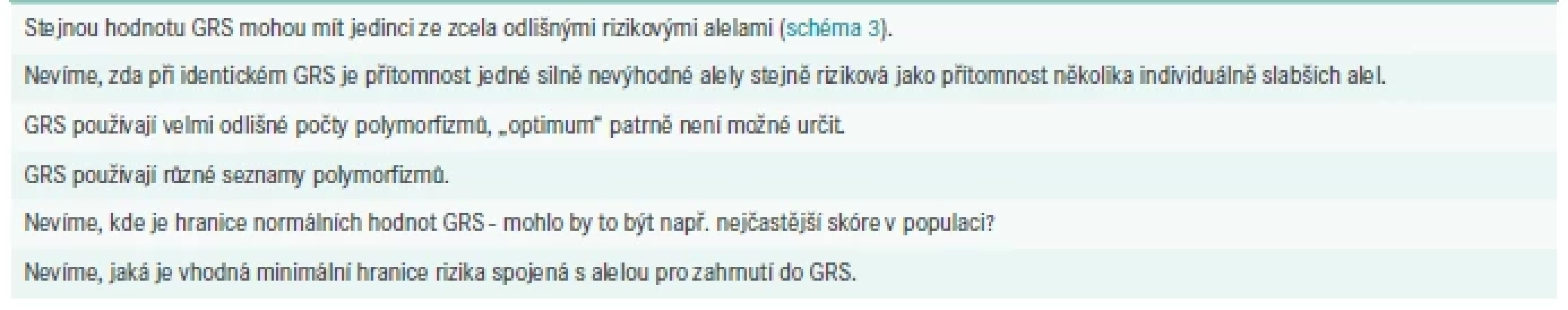

Každá studie je nezbytně limitovaná počtem účastníků a přenos jejích výsledků do individualizované predikce bohužel není tak snadný, jak by se mohlo zdát. Pro správné využití výsledků genetických analýz je tedy třeba replikovat výsledky z původních screeningů na každé dané populaci. Existuje řada variant, které mají etnicky výrazně specifický výskyt i vliv na fenotyp [34]; nicméně význam variant a význam jejich vlivu se může lišit i mezi populacemi stejného etnika. To může být zapříčiněno i interakcí mezi geny a prostředím, výsledky se mohou lišit i mezi muži a ženami. Především nutriční intervence by měly profitovat z implementací výsledků GWAs skrz GRS do individualizovaných doporučení. První studie z této oblasti, kombinující GRS a výživové zvyklosti, jsou slibné [35–37].
Interakce jsou obecně prozatím jednou ze slabin výpočtu GRS/PRS – výpočty počítají s prostým součtem rizik jednotlivých variant. Je ale pravděpodobné, že ve skutečnosti nebude model GRS aditivní a lineární, ale mezi polymorfizmy bude docházet k interakcím [38]. Tedy zjednodušeně, v případě přítomnosti určité kombinace alel se jejich vliv může násobit a efekty jiných se naopak mohou vzájemně vyrušit.
Některé z popsaných problémů se snaží řešit nové techniky výpočtu jako tzv. shared continuous shrinkage [39] nebo transfer learning [40], je však otázkou, zda se opravdu jedná o zjednodušení problematiky PRS nebo zda přílišná depersonalizace konceptu spíše neuškodí.
Dosavadní výsledky ukazují, že GRS jsou důležitějšími prediktory onemocnění v mladším věku a že využití genetických údajů je cennější u subjektů s nižšími riziky prostředí/životního stylu a je platné při reklasifikaci jedinců „na hraně“ z hlediska tradičních klinických RF. Řada studií se ale snaží zjistit (jak je zmíněno výše), co může genetická predisposice přinést ve zlepšení klasifikace rizika nad rámec odhadů vycházející z tradičních rizikových faktorů. Tento přístup je ale v podstatě nesprávný – většina do výpočtů zahrnutých SNPs ovlivňuje právě klinické rizikové faktory, které jsou již u sledovaných pacientů přítomny. GRS predikce by ale ideálně měla identifikovat opravdu mladé jedince ohrožené KVO dříve, než se u nich klinické rizikové faktory vůbec objeví. Nejnovější práce ale ukazují, že PRS je lepším prediktorem KVO než individuální rizikové faktory a signifikantně zlepšuje odhad rizika KVO nad rámec analýz pouze klinických RF.
Autor je podporován projektem MZ ČR – RVO („Institut klinické a experimentální medicíny – IKEM, IČ 00023001“).
Doručeno do redakce 7. 3. 2023
Přijato po recenzi 20. 3. 2023
doc. Ing. Jaroslav A. Hubáček, CSc., DSc.
AtheroRev 2023; 8(2): 95–101
Zdroje
1. Rogers J. The finished genome sequence of Homo sapiens. Cold Spring Harb Symp Quant Biol 2003; 68: 1–11. Dostupné z DOI: <http://dx.doi. org/10.1101/sqb.2003.68.1>.
2. Palomaki GE, Melillo S, Bradley LA. Association between 9p21 genomic markers and heart disease: a meta-analysis. JAMA 2010; 303: 648–656. Dostupné z DOI: <http://dx.doi.org/10.1001/jama.2010.118>.
3. Del Bosque-Plata L, Martínez-Martínez E, Espinoza-Camacho MÁet al. The role of TCF7L2 in type 2 diabetes. Diabetes 2021; 70: 1220–1228. Dostupné z DOI: <http://dx.doi.org/10.2337/db20–0573>.
4. Day FR, Loos RJ. Developments in obesity genetics in the era of genome- wide association studies. J Nutrigenet Nutrigenomics 2011; 4: 222–238. Dostupné z DOI: <http://dx.doi.org/1010.1159/000332158>.
5. Zhao B, Gan L, Graubard BI et al. Associations of dietary cholesterol, serum cholesterol, and egg consumption with overall and cause-specific mortality: Systematic review and updated meta-analysis. Circulation 2022; 145(20): 1506–1520. Dostupné z DOI: <http://dx.doi.org/10.1161/ CIRCULATIONAHA.121.057642>.
6. Zhao Y, Qie R, Han M, et al. Association of BMI with cardiovascular disease incidence and mortality in patients with type 2 diabetes mellitus: A systematic review and dose-response meta-analysis of cohort studies. Nutr Metab Cardiovasc Dis 2021; 31(7): 1976–1984. Dostupné z DOI: <http://dx.doi.org/10.1016/j.numecd.2021.03.003>.
7. Hubáček J, Pistulková H, Valenta Z et al. Genetic predisposition to hypercholesterolemia in childhood. Cor Vasa 1994; 36: 68–72.
8. Valve R, Heikkinen S, Rissanen A et al. Synergistic effect of polymorphisms in uncoupling protein 1 and beta3-adrenergic receptor genes on basal metabolic rate in obese Finns. Diabetologia 1998; 41(3): 357–361. Dostupné z DOI: <http://dx.doi.org/10.1007/s001250050915>.
9. Hubáček JA. Genetické testování pro včasnou predikci kardiovaskulárních onemocnění. Celogenomové asociační studie a polygenní skóre. AtheroRew 2020; 5: 88–92.
10. Janssens ACJW. Validity of polygenic risk scores: are we measuring what we think we are? Hum Mol Genet 2019; 28(R2): R143-R150. Dostupné z DOI: <http://dx.doi.org/10.1093/hmg/ddz205>.
11. Lewis CM, Vassos E. Polygenic risk scores: from research tools to clinical instruments. Genome Med 2020; 12: 44. Dostupné z DOI: <http:// dx.doi.org/10.1186/s13073–020–00742–5>.
12. Igo RP Jr, Kinzy TG, Cooke Bailey JN. Genetic risk scores. Curr Protoc Hum Genet 2019; 104(1): e95. Dostupné z DOI: <http://dx.doi. org/10.1002/cphg.95>.
13. Hubacek JA, Adamkova V, Lanska V, Dlouha D. Polygenic hypercholesterolemia: examples of GWAS results and their replication in the Czech- -Slavonic population. Physiol Res 2017; 66(Suppl 1): S101-S111. Dostupné z DOI: <http://dx.doi.org/10.33549/physiolres.933580>.
14. Lu Y, Feskens EJ, Boer JM et al. Exploring genetic determinants of plasma total cholesterol levels and their predictive value in a longitudinal study. Atherosclerosis 2010; 213(1): 200–205. Dostupné z DOI: <http:// dx.doi.org/10.1016/j.atherosclerosis.2010.08.053>.
15. Shahid SU, Shabana, Cooper JA, et al. Genetic risk analysis of coronary artery disease in Pakistani subjects using a genetic risk score of 21 variants. Atherosclerosis 2017; 258: 1–7. Dostupné z DOI: <http://dx.doi.org/ 10.1016/j.atherosclerosis.2017.01.024>.
16. Shah S, Casas JP, Gaunt TR et al. Influence of common genetic variation on blood lipid levels, cardiovascular risk, and coronary events in two British prospective cohort studies. Eur Heart J 2013; 34: 972–981. Dostupné z DOI: <http://dx.doi.org/10.1093/eurheartj/ehs243>.
17. Wu H, Forgetta V, Zhou S et al. Polygenic risk score for low-density lipoprotein cholesterol is associated with risk of ischemic heart disease and enriches for individuals with familial hypercholesterolemia. Circ Genom Precis Med 2021; 14: e003106. Dostupné z DOI: <http://dx.doi.org/10.1161/ CIRCGEN.120.003106>.
18. Sarraju A, Knowles JW. Genetic testing and risk scores: Impact on familial hypercholesterolemia. Front Cardiovasc Med 2019; 6: 5. Dostupné z DOI: <http://dx.doi.org/10.3389/fcvm.2019.00005>.
19. Vanhoye X, Bardel C, Rimbert A, et al. A new 165-SNP low-density lipoprotein cholesterol polygenic risk score based on next generation sequencing outperforms previously published scores in routine diagnostics of familial hypercholesterolemia. Transl Res 2022; S1931–5244(22)00281-X. Dostupné z DOI: <http://dx.doi.org/10.1016/j.trsl.2022.12.002>.
20. Hubacek JA, Dlouha D, Adamkova V et al. The gene score for predicting hypertriglyceridemia: New insights from a Czech case-control study. Mol Diagn Ther 2019; 23(4): 555–562. Dostupné z DOI: <http://dx.doi. org/10.1007/s40291–019–00412–2>.
21. Johansen CT, Wang J, Lanktree MB, et al. An increased burden of common and rare lipid-associated risk alleles contributes to the phenotypic spectrum of hypertriglyceridemia. Arterioscler Thromb Vasc Biol 2011; 31(8): 1916–1926. Dostupné z DOI: <http://dx.doi.org/10.1161/ ATVBAHA.111.226365>.
22. Fava C, Sjögren M, Montagnana M et al. Prediction of blood pressure changes over time and incidence of hypertension by a genetic risk score in Swedes. Hypertension 2013; 61(2): 319–326. Dostupné z DOI: <http:// dx.doi.org/10.1161/HYPERTENSIONAHA.112.202655>.
23. Damavandi N, Soleymaniniya A, Bahrami Zadegan S et al. Development of a genetic risk score for obesity predisposition evaluation. Mol Genet Genomics 2022; 297(6): 1495–1503. Dostupné z DOI: <http://dx.doi. org/10.1007/s00438–022–01923–0>.
24. Talmud PJ, Cooper JA, Morris RW et al. Sixty-five common genetic variants and prediction of type 2 diabetes. Diabetes 2015; 64(5): 1830–40. Dostupné z DOI: <http://dx.doi.org/10.2337/db14–1504>.
25. Zarkoob H, Lewinsky S, Almgren P et al. Utilization of genetic data can improve the prediction of type 2 diabetes incidence in a Swedish cohort. PLoS One 2017; 12(7): e0180180. Dostupné z DOI: <http://dx.doi. org/10.1371/journal.pone.0180180>.
26. Goodarzi MO, Palmer ND, Cui J et al., Classification of type 2 diabetes genetic variants and a novel genetic risk score association with insulin clearance. J Clin Endocrinol Metab 2020; 105(4): 1251–1260. Dostupné z DOI: <http://dx.doi.org/10.1210/clinem/dgz198>.
27. Yun H, Noh NI Lee EY. Genetic risk scores used in cardiovascular disease prediction models: a systematic review. Rev Cardiovasc Med 2022; 23(1): 8. Dostupné z DOI: <http://dx.doi.org/10.31083/j.rcm2301008>.
28. Thériault S, Lali R, Chong M et al. Polygenic contribution in individuals with early-onset coronary artery disease. Circ Genom Precis Med 2018; 11(1): e001849. Dostupné z DOI: <http://dx.doi.org/10.1161/ CIRCGEN.117.001849>.
29. Khera AV, Chaffin M, Zekavat SM et al. Whole-genome sequencing to characterize monogenic and polygenic contributions in patients hospitalized with early-onset myocardial infarction. Circulation 2019; 139: 1593–1602. Dostupné z DOI: <http://dx.doi.org/10.1161/ CIRCULATIONAHA.118.035658>.
30. Krarup NT, Borglykke A, Allin KH, et al. A genetic risk score of 45 coronary artery disease risk variants associates with increased risk of myocardial infarction in 6041 Danish individuals. Atherosclerosis 2015; 240(2): 305–310. Dostupné z DOI: <http://dx.doi.org/10.1016/j. atherosclerosis.2015.03.022>.
31. Morris RW, Cooper JA, Shah T et al. Marginal role for 53 common genetic variants in cardiovascular disease prediction. Heart 2016; 102: 1640–1647. Dostupné z DOI: <http://dx.doi.org/10.1136/heartjnl-2016–309298>.
32. Meigs JB, Shrader P, Sullivan LM, et al. Genotype score in addition to common risk factors for prediction of type 2 diabetes. N Engl J Med 2008; 359(21): 2208–2219. Dostupné z DOI: <http://dx.doi.org/10.1056/ NEJMoa0804742. Erratum in: N Engl J Med 2009; 360(6): 648>.
33. Mosley JD, Gupta DK, Tan J et al. Predictive accuracy of a polygenic risk score compared with a clinical risk score for incident coronary heart disease. JAMA 2020; 323(7): 627–635. Dostupné z DOI: <http://dx.doi. org/10.1001/jama.2019.21782>.
34. Huang T, Shu Y, Cai YD. Genetic differences among ethnic groups. BMC Genomics 2015; 16: 1093. Dostupné z DOI: <http://dx.doi.org/10.1186/ s12864–015–2328–0>.
35. Whayne TF Jr, Saha SP. Genetic risk, adherence to a healthy lifestyle, and ischemic heart disease. Curr Cardiol Rep 2019; 21(1): 1. Dostupné z DOI: <http://dx.doi.org/10.1007/s11886–019–1086-z>.
36. Park SJ, Kim MS, Choi SW, Lee HJ. The relationship of dietary pattern and genetic risk score with the incidence dyslipidemia: 14-year follow- -up cohort study. Nutrients 2020; 12(12): 3840. Dostupné z DOI: <http:// dx.doi.org/10.3390/nu12123840>.
37. Pang S, Yengo L, Nelson CP et al. Genetic and modifiable risk factors combine multiplicatively in common disease. Clin Res Cardiol 2023; 112(2): 247–257. Dostupné z DOI: <http://dx.doi.org/10.1007/ s00392–022–02081–4>.
38. Hubáček JA, Vrablík M. Genetika dyslipidemií včera, dnes a zítra. Vnitř Lék 2007; 53(4): 371–376.
39. Ruan Y, Lin YF, Feng YA et al. Improving polygenic prediction in ancestrally diverse populations. Nat Genet 2022; 54(5): 573–580. Dostupné z DOI: <http://dx.doi.org/doi:10.1038/s41588–022–01054–7>.
40. Zhao Z, Fritsche LG, Smith JA et al. The construction of cross-population polygenic risk scores using transfer learning. Am J Hum Genet 2022; 109(11): 1998–2008. Dostupné z DOI: <http://dx.doi.org/10.1016/j. ajhg.2022.09.010>.
Štítky
Angiologie Diabetologie Interní lékařství Kardiologie Praktické lékařství pro dospěléČlánek vyšel v časopise
Athero Review

2023 Číslo 2
Nejčtenější v tomto čísle
- Kyselina bempedoová v kontextu současných možností hypolipidemické léčby a výsledků studie CLEAR OUTCOMES
- Stanovisko ČSAT ke Konsenzu Evropské společnosti pro aterosklerózu: Lipoprotein(a) při aterosklerotických kardiovaskulárních onemocněních a aortální stenóze
- Význam nestatínových liekov pre znižovanie LDL-cholesterolu v manažmente veľmi vysokého a vysokého aterosklerotického kardiovaskulárneho rizika
- Hyperlipidemická krize jako rizikový faktor pro vznik akutní pankreatitidy
